之前学习了几种分类的算法:逻辑回归、KNN近邻。这次来看另外一种模型:决策树。决策树是一种对实例进行分类的树形结构,通过多层判断区分目标所属类别。决策树的本质就是通过多层判断,从训练数据集中归纳出一组分类规则。其优点:计算量小,运算速度快;易于理解,可清晰查看各种属性的重要性。但是容易忽略属性间的相关性;样本类别分布不均匀时,容易影响模型表现。
<!-- more --> <!--more--> 一个简单的例子:根据用户的学习动力、能力提升意愿、兴趣度、空余时间,判断其是否适合学习本门课程。
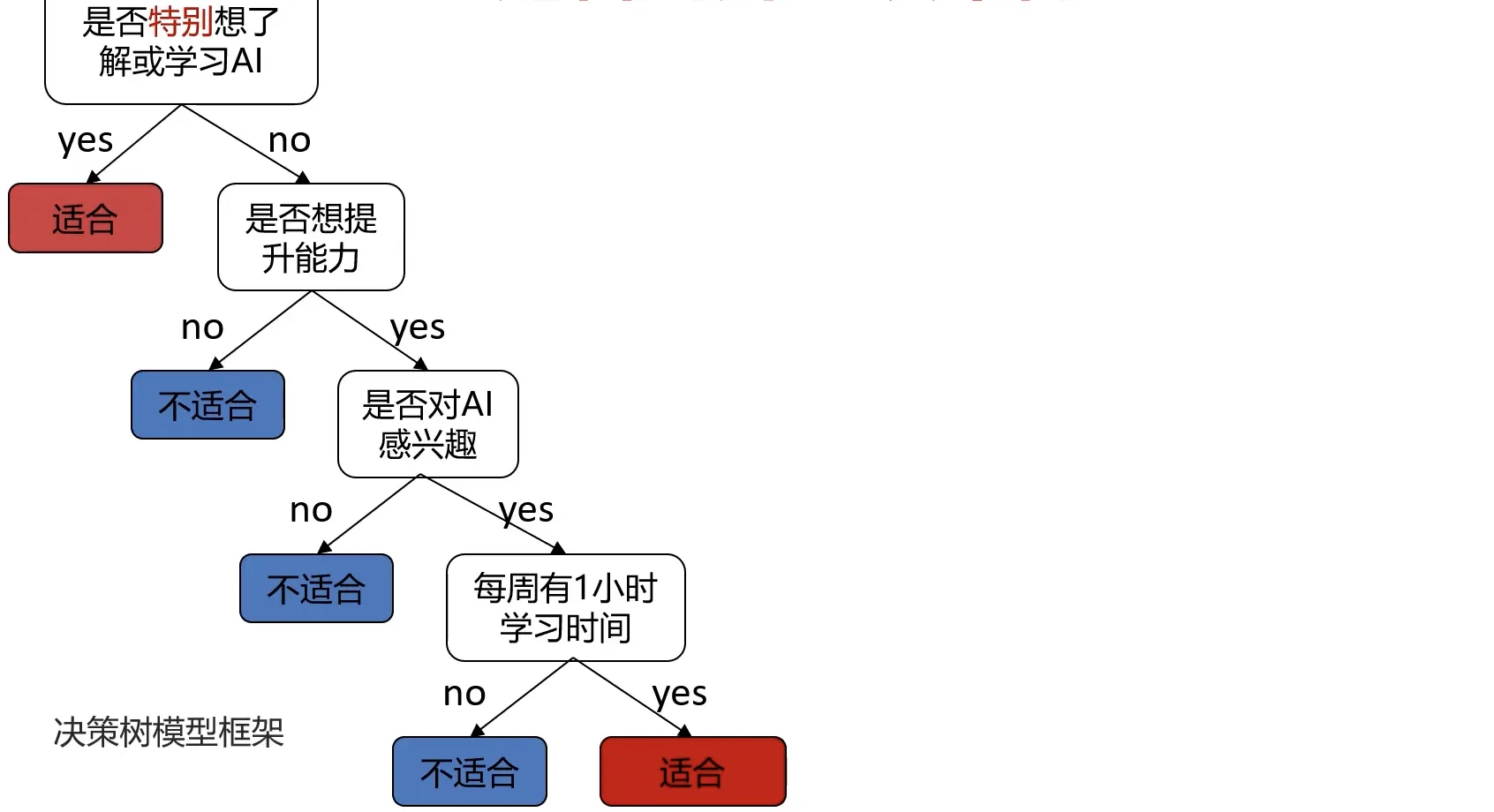
那么问题来了,凭啥“是否特别想了解或学习AI” 是首要判断条件呢?这就涉及到决策树求解的过程了。
决策树(DecisionTree)
假设给定训练数据集 $D={(x_1,y_1),(x_2,y_2),\ldots,(xN,yN)}$ ,其中 $x_i=(x_i^{(1)},x_i^{(2)},\ldots,x_i^{(m)})^T$ 为输入实例,m 为特征个数,$y_i\in{1,2,3,\ldots,K}$ 为类标记,$i = 1,2,……N$, N 为样本容量。
目标:根据训练数据集构建一个决策树模型,使它能够对实例进行正确的分类。
问题核心:特征选择,每一个节点选哪一个特征。选择的特征不同,决策树也不同,那么选择哪一个特征的决策树呢?有三种方法:ID3,C4.5,CART(前两者用到了信息增益的概念)
这里不讨论复杂的数学公式推导,简单来看,ID3:利用信息熵原理选择信息增益最大的属性作为分类属性,递归地拓展决策树的分支,完成决策树的构造。
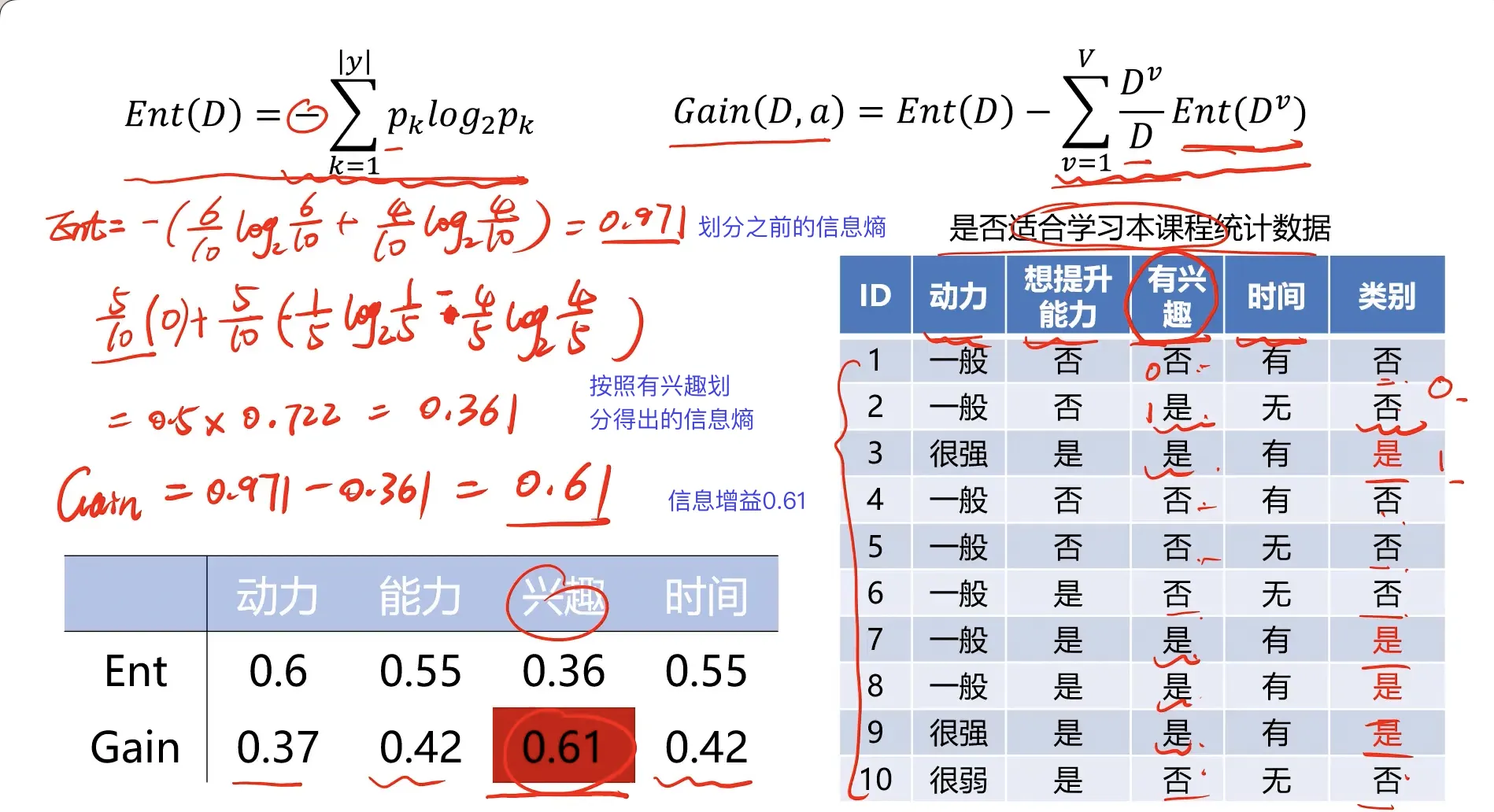
信息熵的定义:是度量随机变量不确定性的指标,熵越大,变量的不确定性越大。假定当前样本集合D中的第k类样本所占比例为 $p_k$ ,则D的信息熵为: $$ Ent(D)=-\sum_{k=1}^{|y|}p_klog_2p_k $$
Ent(D) 的值越小,变量的不确定性越小。$p_k = 1$ 时,$Ent(D)= 0$,$p_k = 0$ 时,$Ent(D)= 0$。因为两种情况都表示另外一种情况为不可能,所以确定性是100%。
根据信息熵,可以计算以属性a进行样本划分带来的信息增益:
$$Gain(D,a)=Ent(D)-\sum_{v=1}^{V}\frac{D^v}DEnt(D^v)$$
V为根据属性 a 划分出的类别数、D为当前样本总数,$D^v$ 为类别V的样本数。
$Ent(D)$ 就是划分之前的信息熵,$\sum_{v=1}^{V}\frac{D^v}DEnt(D^v)$ 就是划分之后的信息熵。
目标: 划分后样本分布不确定性尽可能小,即划分后信息熵小,信息增益大。因为你肯定希望每次划分完毕之后的不确定性逐渐变小的。
以上面的例子来计算一次:
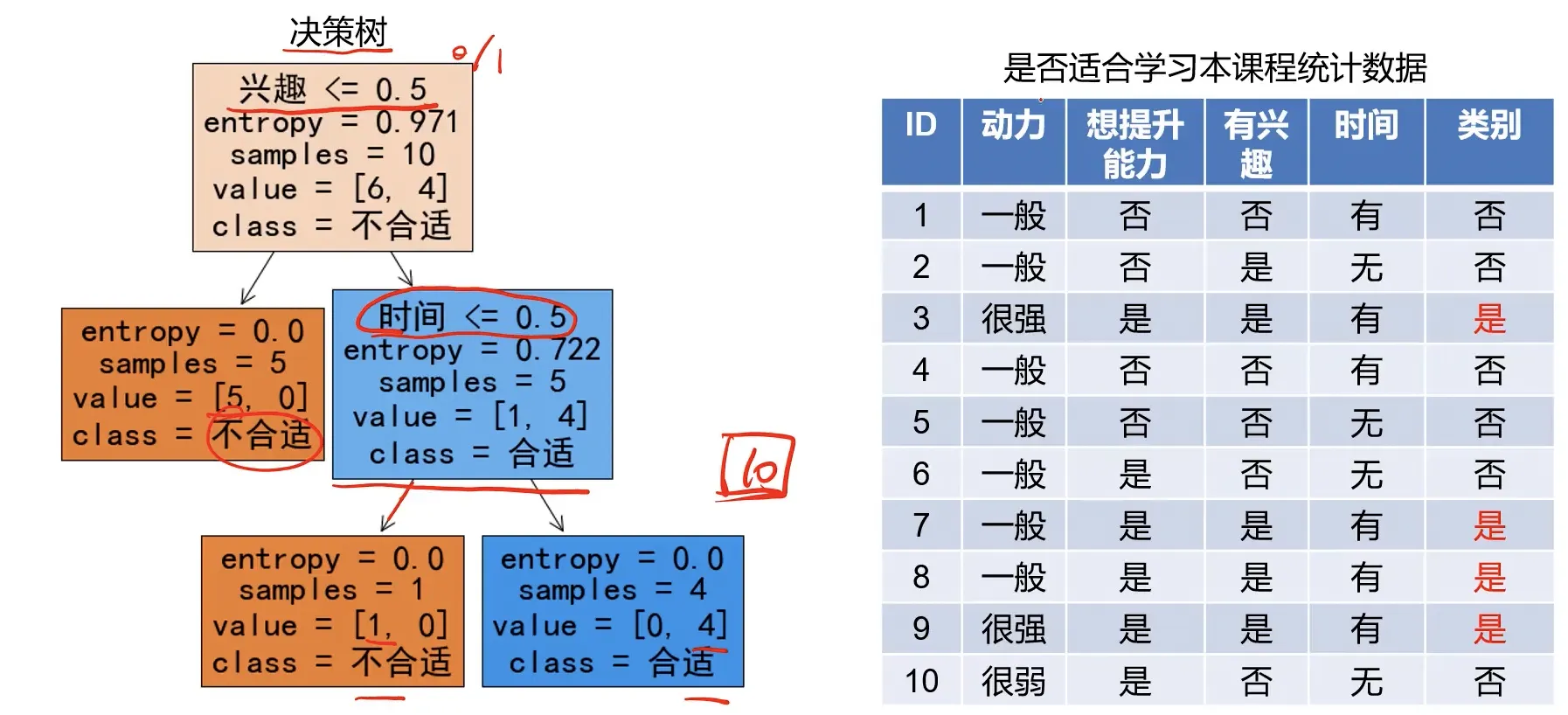
经过计算可以得到如下的决策树:
异常检测(Anomaly Detection)
异常检测其实就是根据输入数据,对不符合预期模式的数据进行识别:

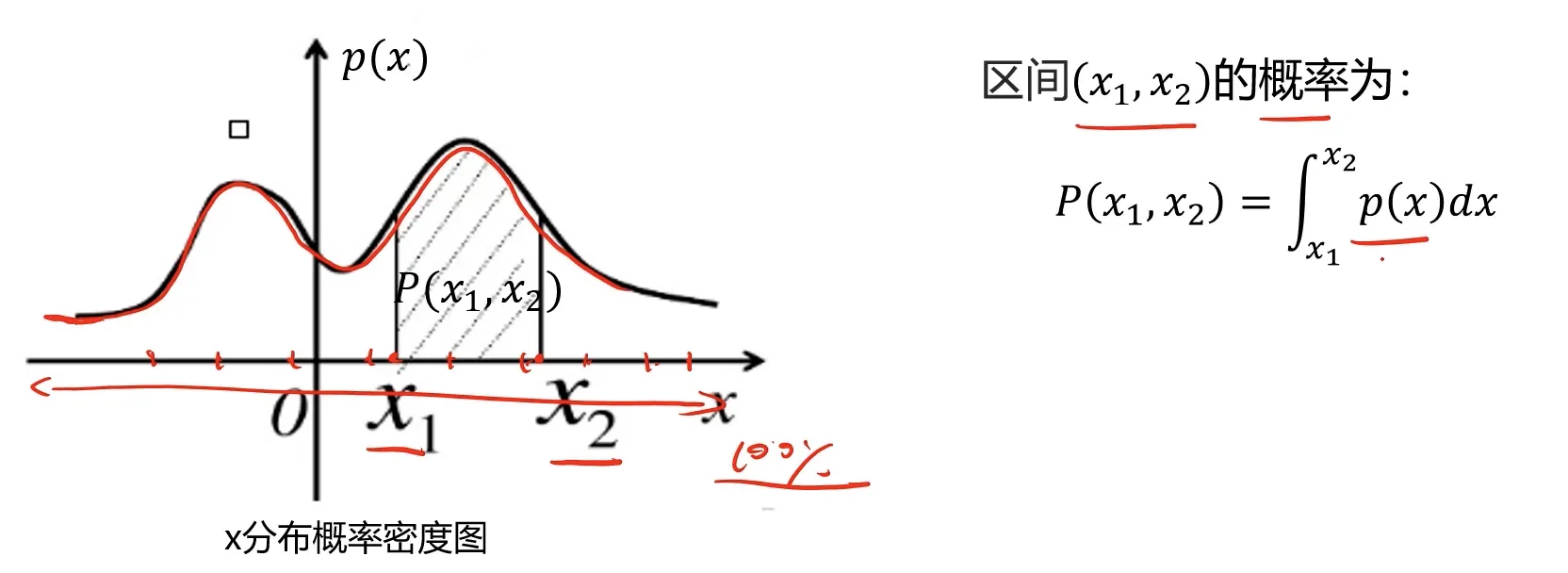
这里我们需要引入一个概率密度的概念:概率密度函数是一个描述随机变量在某个确定的取值点附近为可能性的函数。
通常情况下,数据的概率密度是符合高斯分布的。高斯分布的概率密度函数是(这些是概率论的基础内容):
$$p(x)=\frac1{\sigma\sqrt{2\pi}}e^{-\frac{(x-\mu)^2}{2\sigma^2}}$$
其中μ为数据均值,σ为标准差:
$$\begin{gathered}\mu=\frac1m\sum_{i=1}^{m}x^{(i)},\\sigma^2=\frac1m\sum_{i=1}^{m}(x^{(i)}-\mu)^2\end{gathered}$$
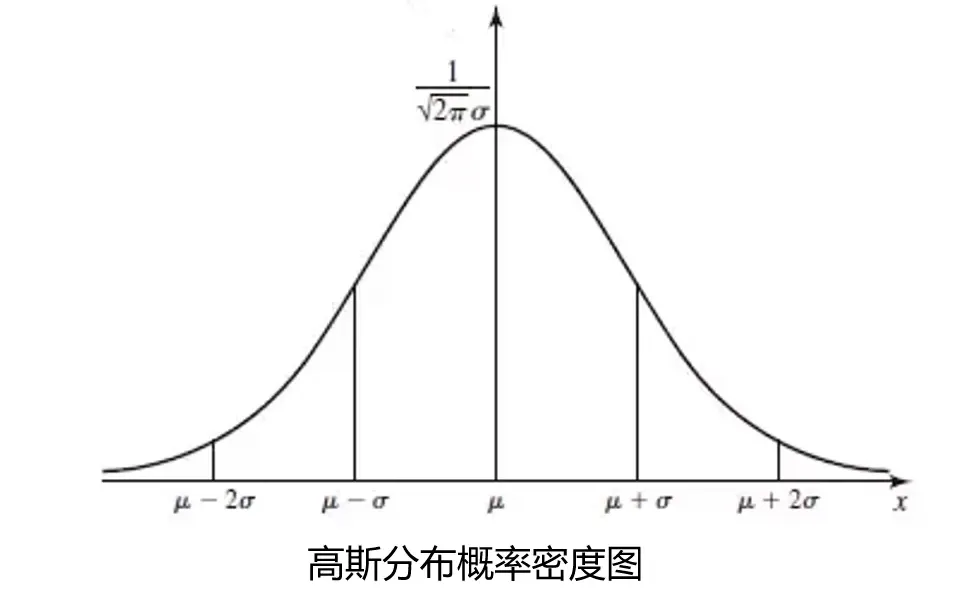
一维数据基于高斯分布实现异常检测:
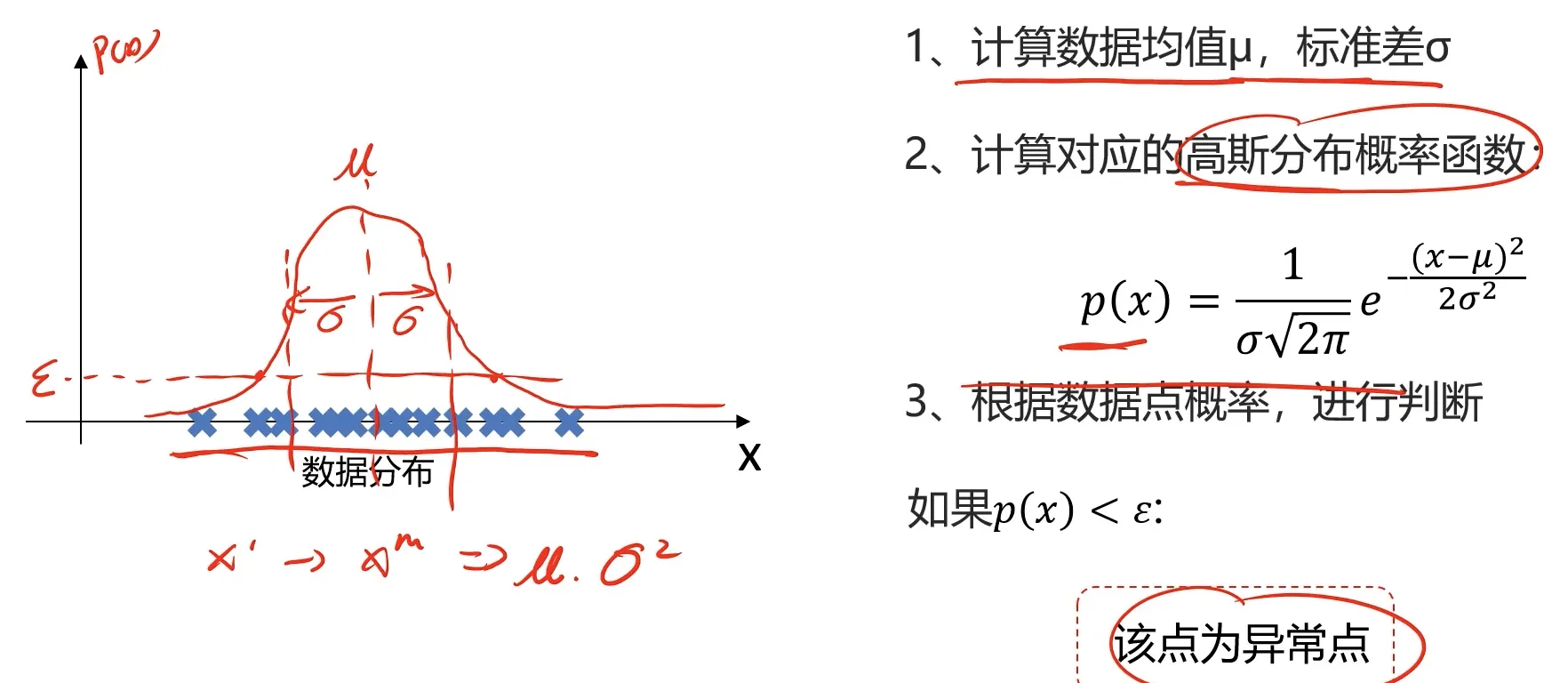
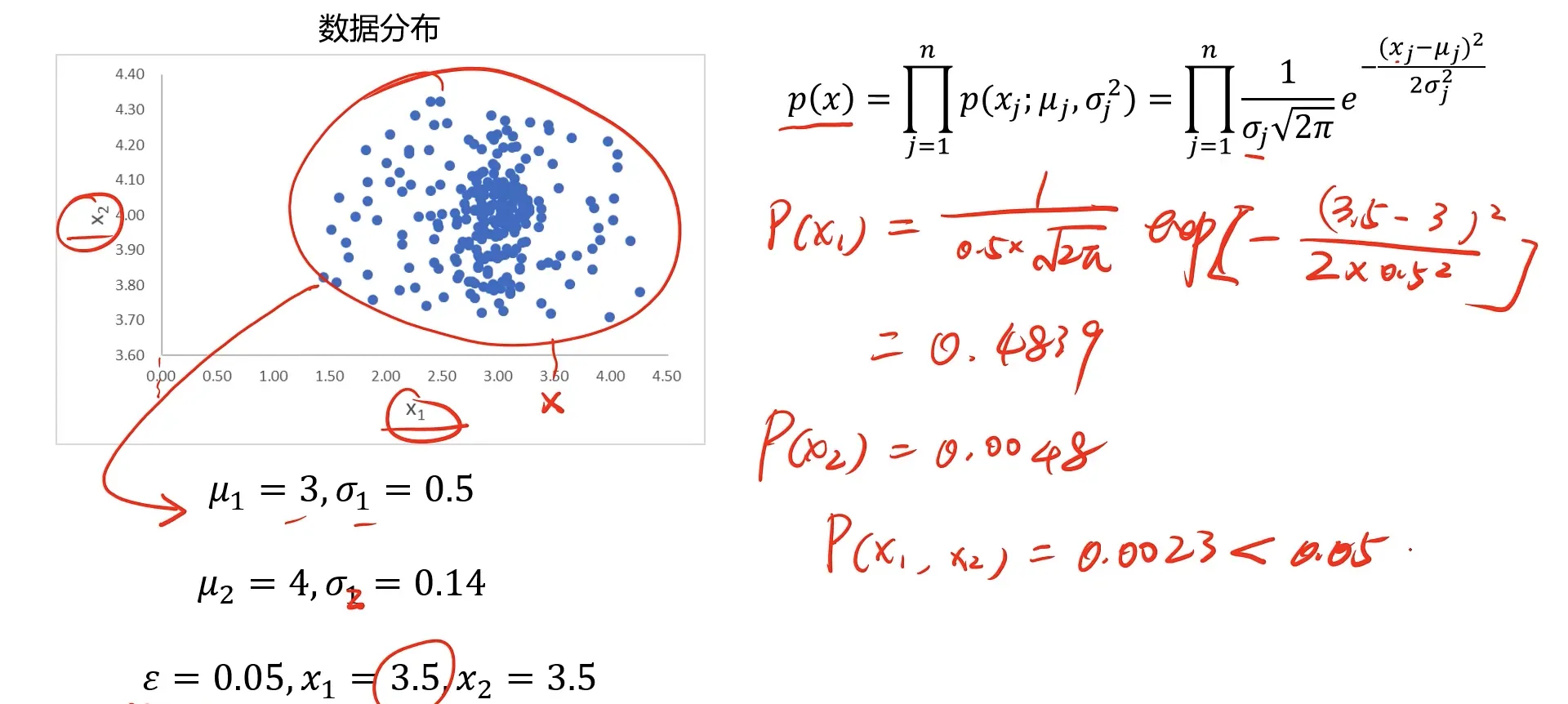
N维数据基于高斯分布实现异常检测:
$$\begin{Bmatrix}{x_1^{(1)},x_1^{(2)},...x_1^{(m)}}_{...}\...\x_n^{(1)},x_n^{(2)},...x_n^{(m)}\end{Bmatrix}$$
对于N维的情况我们可以先计算每一个维度下的均值和标准差:
$$\mu_j=\frac1m\sum_{i=1}^mx_j^{(i)},\quad\quad\sigma_j^2=\frac1m\sum_{i=1}^m(x_j^{(i)}-\mu_j)^2$$
然后得出对应维度下的概率密度函数,再把每个维度下的概率密度函数相乘。
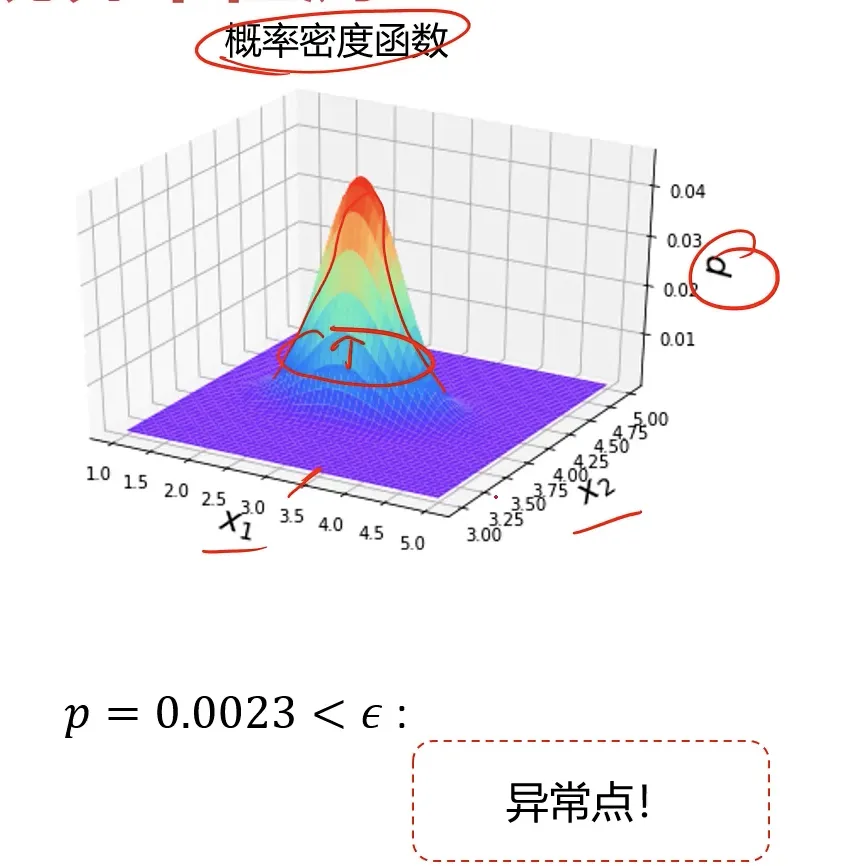
$$\begin{aligned}{p(x)}&=\prod_{j=1}^np(x_j;\mu_j,\sigma_j^2)=\prod_{j=1}^n\frac{1}{\sigma_j\sqrt{2\pi}}e^{-\frac{(x_j-\mu_j)^2}{2\sigma_j^2}}\end{aligned}$$
通过可视化的方式看起来是这样:
主成分分析(PCA-Principal component analysis)
有一个数据降维的真实案例: 通过美国1929-1938年各年经济数据,预测国民收入与支出。数据包括:雇主补贴、消费资料和生产资料、纯公共支出、净增库存、股息、利息、外贸平衡等十七个指标。一个叫做斯通(Stone)的统计学家提出了一个方法:主成分分析。将17个指标的模型降到了3个指标(这里三个指标并不是初始的三个指标),预测准确率达到97%。
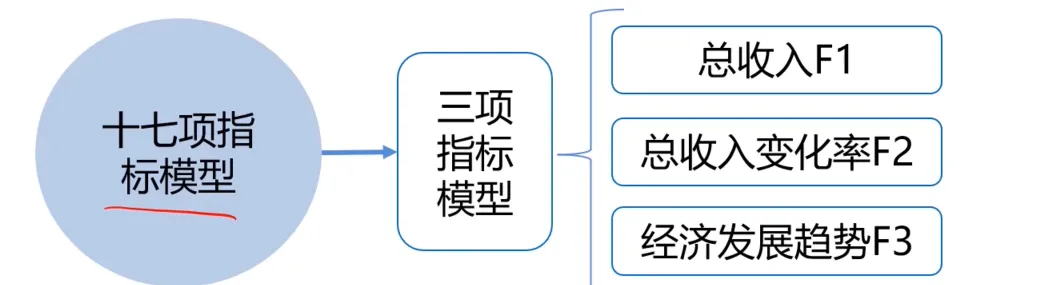
数据降维,是指在某些限定条件下降低随机变量个数得到一组“不相关”主变量的过程。
作用:1、减少模型分析数据量,提升处理效率,降低计算难度2、实现数据可视化。
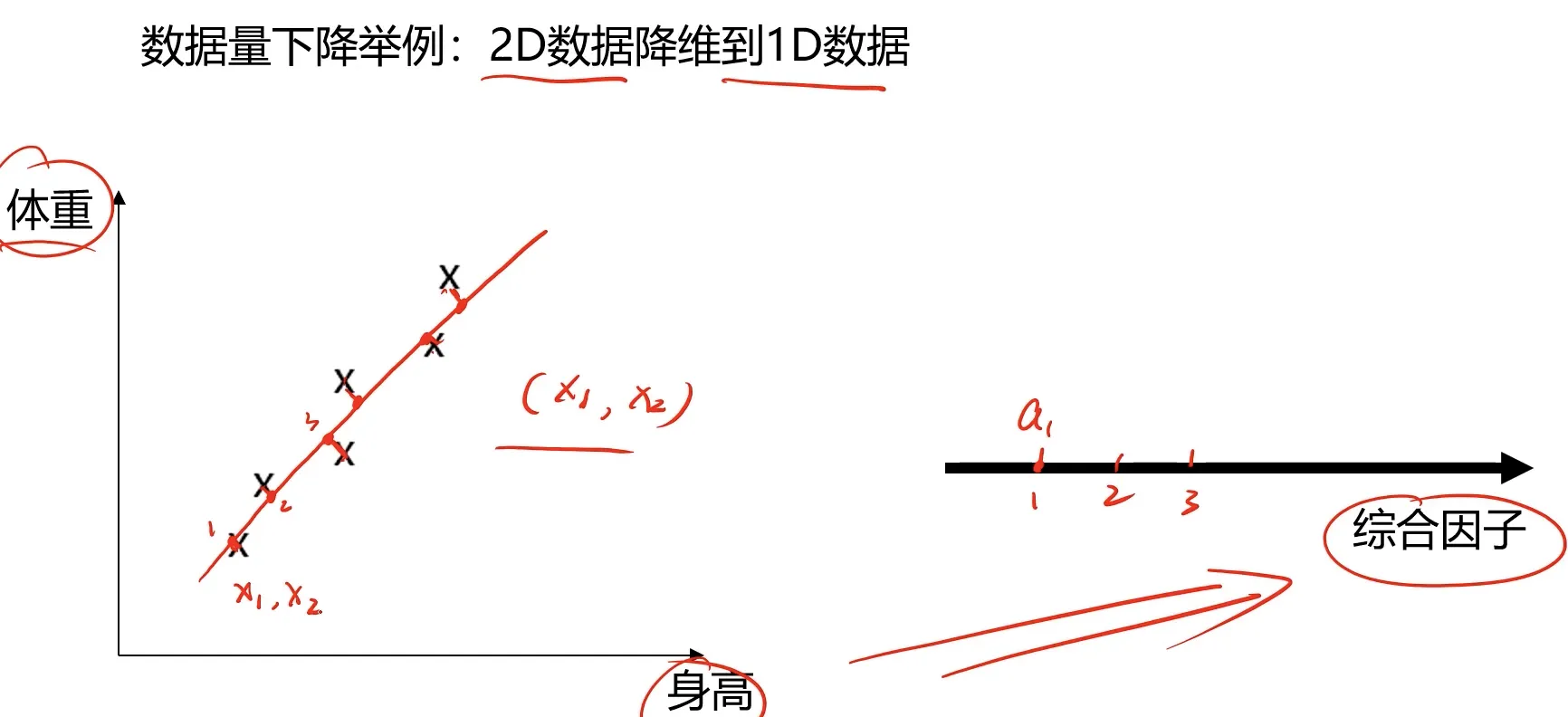
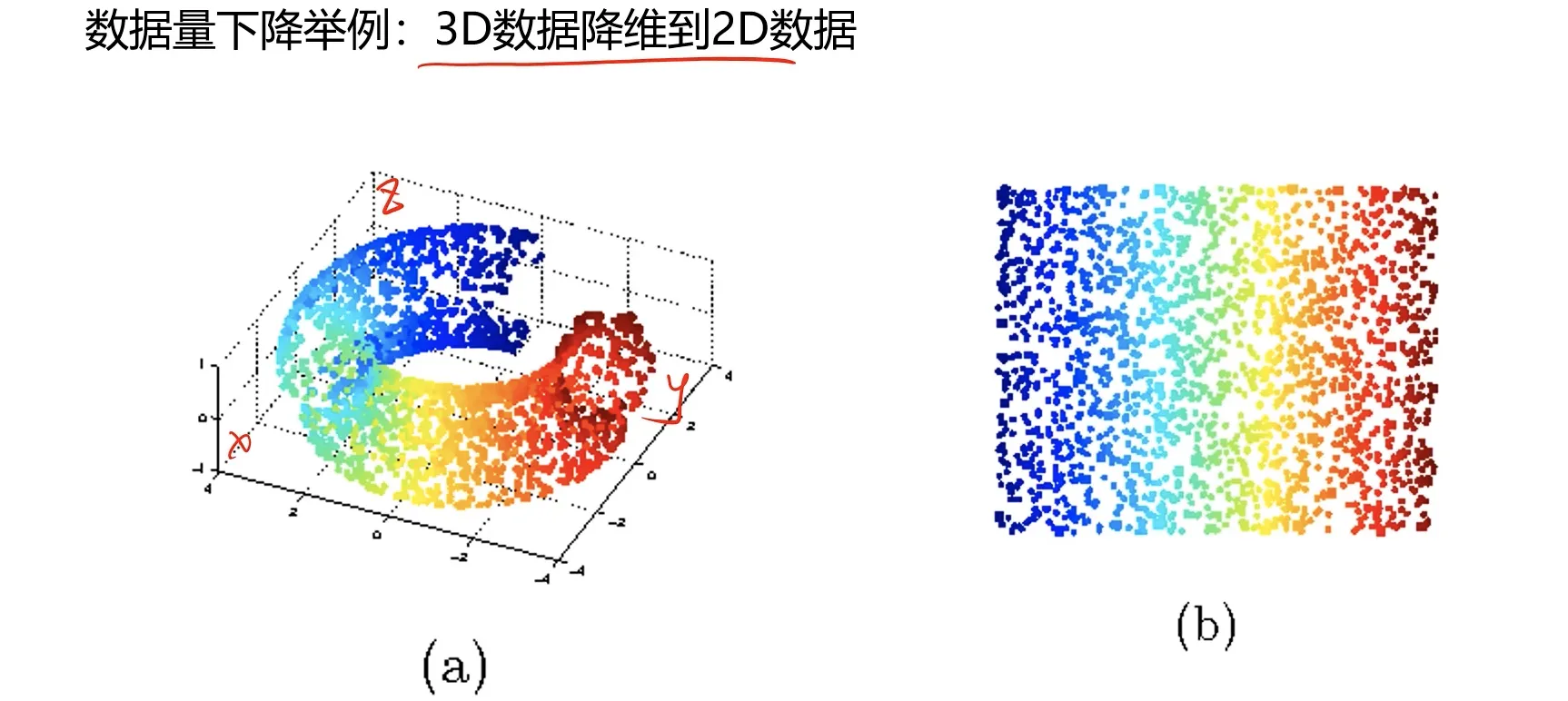
主成分分析(PCA)作为数据降维的实现,也是应用最多的方法,其目标就是寻找k(k<n)维新数据,使他们反应事物的主要特征。
核心:在信息损失尽可能少的情况下,降低数据维度
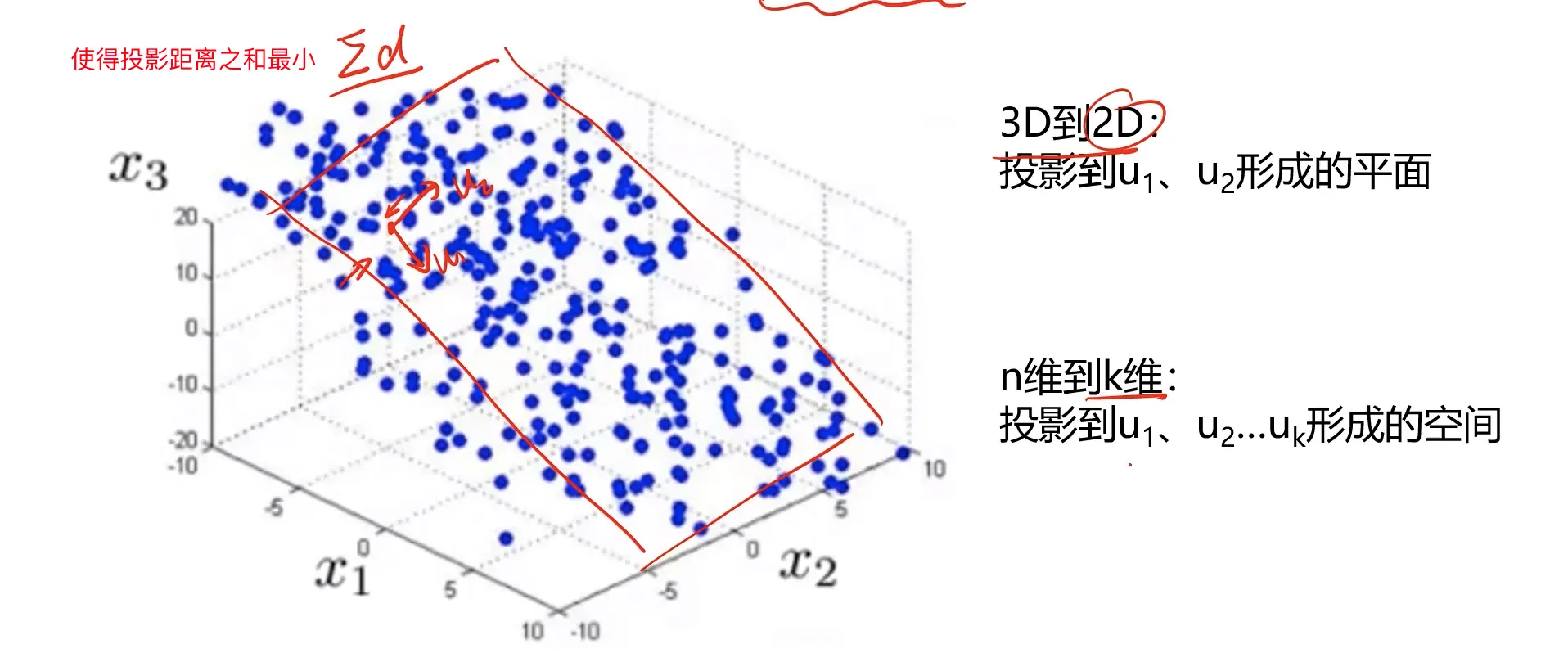
如何保留主要信息:投影后的不同特征数据尽可能分得开(即不相关)
如何实现:使投影后数据的方差最大,因为方差越大数据也越分散
计算过程:
- 1、原始数据预处理(标准化:μ=0, σ=1)
- 2、计算协方差矩阵特征向量,及数据在各特征向量投影后的方差
- 3、根据需求(任务指定或方差比例)确定降维维度k
- 4、选取k维特征向量,计算数据在其形成空间的投影
决策树案例实战
lIris数据集:lris 尾花数据集是一个经典数据集,在统计学习和机器学习领域都经常被用作示例。作为机器学习经典数据集:简单而具有代表性,常用于监督式学习应用。

3共150条记录,每类各 50个数据。每条记录都有4项特:征花萼长度(Sepal Length)、花萼宽度(Sepal Width)、花瓣长度(Petal Length)、花瓣宽度(Petal Width)。
目标呢就是通过4个特征预测花卉属于三类 (iris-setosa, iris-versicolour, iris-virginica)中的哪一品种。

决策树实战task:
1、基于iris_data.csv数据,建立决策树模型,评估模型表现
2、可视化决策树结构
3、修改min_samples_leaf参数,对比模型结果
import pandas as pd
import numpy as np
data = pd.read_csv('iris_data.csv')
data.head()
<table border="1" class="dataframe"> <thead> <tr style="text-align: right;"> <th></th> <th>sepal length</th> <th>sepal width</th> <th>petal length</th> <th>petal width</th> <th>target</th> <th>label</th> </tr> </thead> <tbody> <tr> <th>0</th> <td>5.1</td> <td>3.5</td> <td>1.4</td> <td>0.2</td> <td>Iris-setosa</td> <td>0</td> </tr> <tr> <th>1</th> <td>4.9</td> <td>3.0</td> <td>1.4</td> <td>0.2</td> <td>Iris-setosa</td> <td>0</td> </tr> <tr> <th>2</th> <td>4.7</td> <td>3.2</td> <td>1.3</td> <td>0.2</td> <td>Iris-setosa</td> <td>0</td> </tr> <tr> <th>3</th> <td>4.6</td> <td>3.1</td> <td>1.5</td> <td>0.2</td> <td>Iris-setosa</td> <td>0</td> </tr> <tr> <th>4</th> <td>5.0</td> <td>3.6</td> <td>1.4</td> <td>0.2</td> <td>Iris-setosa</td> <td>0</td> </tr> </tbody> </table> </div>
#define the X and y
X = data.drop(['target', 'label'],axis=1)
y = data.loc[:, 'label']
print(X.shape, y.shape)
(150, 4) (150,)
#建立决策树模型
from sklearn import tree
dc_tree = tree.DecisionTreeClassifier(criterion='entropy', min_samples_leaf=5)
dc_tree.fit(X,y)
| DecisionTreeClassifier(criterion='entropy', min_samples_leaf=5) |
|---|
#评估模型表现
y_predict = dc_tree.predict(X)
from sklearn.metrics import accuracy_score
accuracy = accuracy_score(y, y_predict)
print(accuracy)
0.9733333333333334
# 可视化展示
from matplotlib import pyplot as plt
fig1 = plt.figure(figsize=(10,10))
#tree.plot_tree(dc_tree)
# 加上分类名称
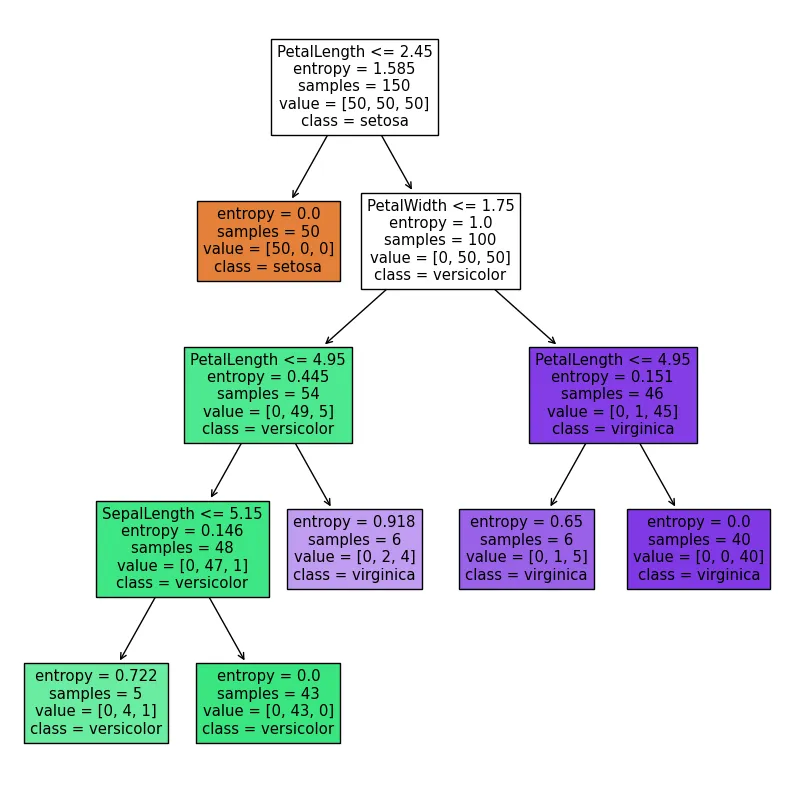
tree.plot_tree(dc_tree,filled=True,feature_names=['SepalLength', 'SepalWidth', 'PetalLength', 'PetalWidth'],class_names=['setosa','versicolor','virginica'])
[Text(0.4444444444444444, 0.9, 'PetalLength <= 2.45\nentropy = 1.585\nsamples = 150\nvalue = [50, 50, 50]\nclass = setosa'),
Text(0.3333333333333333, 0.7, 'entropy = 0.0\nsamples = 50\nvalue = [50, 0, 0]\nclass = setosa'),
Text(0.5555555555555556, 0.7, 'PetalWidth <= 1.75\nentropy = 1.0\nsamples = 100\nvalue = [0, 50, 50]\nclass = versicolor'),
Text(0.3333333333333333, 0.5, 'PetalLength <= 4.95\nentropy = 0.445\nsamples = 54\nvalue = [0, 49, 5]\nclass = versicolor'),
Text(0.2222222222222222, 0.3, 'SepalLength <= 5.15\nentropy = 0.146\nsamples = 48\nvalue = [0, 47, 1]\nclass = versicolor'),
Text(0.1111111111111111, 0.1, 'entropy = 0.722\nsamples = 5\nvalue = [0, 4, 1]\nclass = versicolor'),
Text(0.3333333333333333, 0.1, 'entropy = 0.0\nsamples = 43\nvalue = [0, 43, 0]\nclass = versicolor'),
Text(0.4444444444444444, 0.3, 'entropy = 0.918\nsamples = 6\nvalue = [0, 2, 4]\nclass = virginica'),
Text(0.7777777777777778, 0.5, 'PetalLength <= 4.95\nentropy = 0.151\nsamples = 46\nvalue = [0, 1, 45]\nclass = virginica'),
Text(0.6666666666666666, 0.3, 'entropy = 0.65\nsamples = 6\nvalue = [0, 1, 5]\nclass = virginica'),
Text(0.8888888888888888, 0.3, 'entropy = 0.0\nsamples = 40\nvalue = [0, 0, 40]\nclass = virginica')]
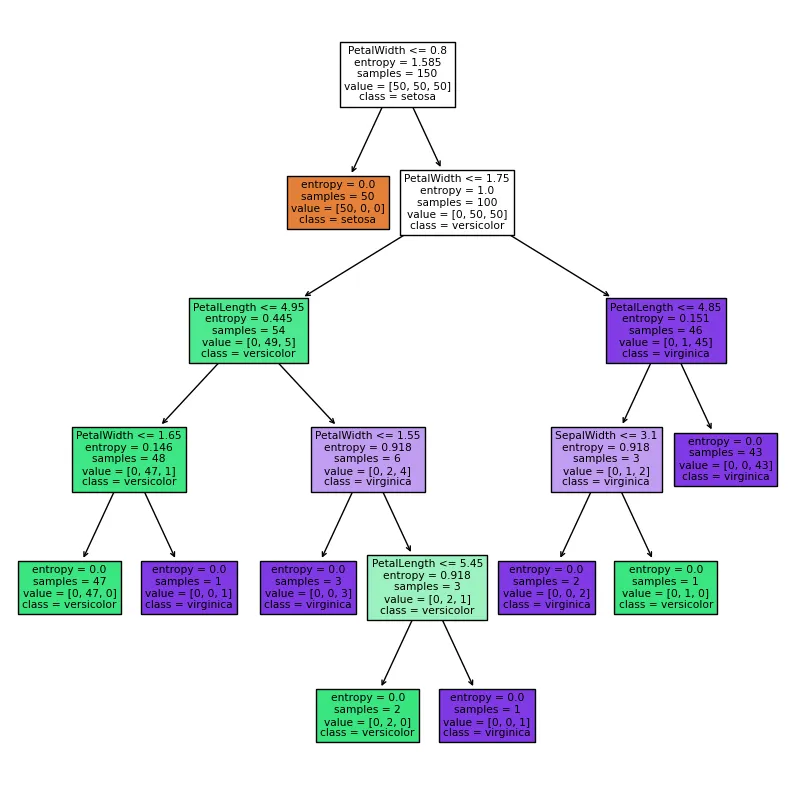
dc_tree = tree.DecisionTreeClassifier(criterion='entropy', min_samples_leaf=1)
dc_tree.fit(X,y)
fig2 = plt.figure(figsize=(10,10))
# 加上分类名称
tree.plot_tree(dc_tree,filled=True,feature_names=['SepalLength', 'SepalWidth', 'PetalLength', 'PetalWidth'],class_names=['setosa','versicolor','virginica'])
[Text(0.5, 0.9166666666666666, 'PetalWidth <= 0.8\nentropy = 1.585\nsamples = 150\nvalue = [50, 50, 50]\nclass = setosa'),
Text(0.4230769230769231, 0.75, 'entropy = 0.0\nsamples = 50\nvalue = [50, 0, 0]\nclass = setosa'),
Text(0.5769230769230769, 0.75, 'PetalWidth <= 1.75\nentropy = 1.0\nsamples = 100\nvalue = [0, 50, 50]\nclass = versicolor'),
Text(0.3076923076923077, 0.5833333333333334, 'PetalLength <= 4.95\nentropy = 0.445\nsamples = 54\nvalue = [0, 49, 5]\nclass = versicolor'),
Text(0.15384615384615385, 0.4166666666666667, 'PetalWidth <= 1.65\nentropy = 0.146\nsamples = 48\nvalue = [0, 47, 1]\nclass = versicolor'),
Text(0.07692307692307693, 0.25, 'entropy = 0.0\nsamples = 47\nvalue = [0, 47, 0]\nclass = versicolor'),
Text(0.23076923076923078, 0.25, 'entropy = 0.0\nsamples = 1\nvalue = [0, 0, 1]\nclass = virginica'),
Text(0.46153846153846156, 0.4166666666666667, 'PetalWidth <= 1.55\nentropy = 0.918\nsamples = 6\nvalue = [0, 2, 4]\nclass = virginica'),
Text(0.38461538461538464, 0.25, 'entropy = 0.0\nsamples = 3\nvalue = [0, 0, 3]\nclass = virginica'),
Text(0.5384615384615384, 0.25, 'PetalLength <= 5.45\nentropy = 0.918\nsamples = 3\nvalue = [0, 2, 1]\nclass = versicolor'),
Text(0.46153846153846156, 0.08333333333333333, 'entropy = 0.0\nsamples = 2\nvalue = [0, 2, 0]\nclass = versicolor'),
Text(0.6153846153846154, 0.08333333333333333, 'entropy = 0.0\nsamples = 1\nvalue = [0, 0, 1]\nclass = virginica'),
Text(0.8461538461538461, 0.5833333333333334, 'PetalLength <= 4.85\nentropy = 0.151\nsamples = 46\nvalue = [0, 1, 45]\nclass = virginica'),
Text(0.7692307692307693, 0.4166666666666667, 'SepalWidth <= 3.1\nentropy = 0.918\nsamples = 3\nvalue = [0, 1, 2]\nclass = virginica'),
Text(0.6923076923076923, 0.25, 'entropy = 0.0\nsamples = 2\nvalue = [0, 0, 2]\nclass = virginica'),
Text(0.8461538461538461, 0.25, 'entropy = 0.0\nsamples = 1\nvalue = [0, 1, 0]\nclass = versicolor'),
Text(0.9230769230769231, 0.4166666666666667, 'entropy = 0.0\nsamples = 43\nvalue = [0, 0, 43]\nclass = virginica')]
异常检测实战
异常检测实战task:
1、基于 anomaly_data.csv数据,可视化数据分布情况、及其对应高斯分布的概率密度函数
2、建立模型,实现异常数据点预测
3、可视化异常检测处理结果
4、修改概率分布阈值EllipticEnvelope(contamination=0.1)中的contamination,查看阈值改变对结果的影响
#load the data
import pandas as pd
import numpy as np
data = pd.read_csv('anomaly_data.csv')
data.head()
<table border="1" class="dataframe"> <thead> <tr style="text-align: right;"> <th></th> <th>x1</th> <th>x2</th> </tr> </thead> <tbody> <tr> <th>0</th> <td>8.046815</td> <td>9.741152</td> </tr> <tr> <th>1</th> <td>8.408520</td> <td>8.763270</td> </tr> <tr> <th>2</th> <td>9.195915</td> <td>10.853181</td> </tr> <tr> <th>3</th> <td>9.914701</td> <td>11.174260</td> </tr> <tr> <th>4</th> <td>8.576700</td> <td>9.042849</td> </tr> </tbody> </table> </div>
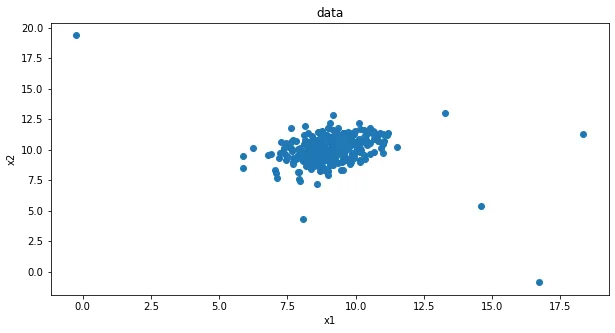
#visualize the data
%matplotlib inline
from matplotlib import pyplot as plt
fig1 = plt.figure(figsize=(10,5))
plt.scatter(data.loc[:,'x1'],data.loc[:,'x2'])
plt.title('data')
plt.xlabel('x1')
plt.ylabel('x2')
plt.show()
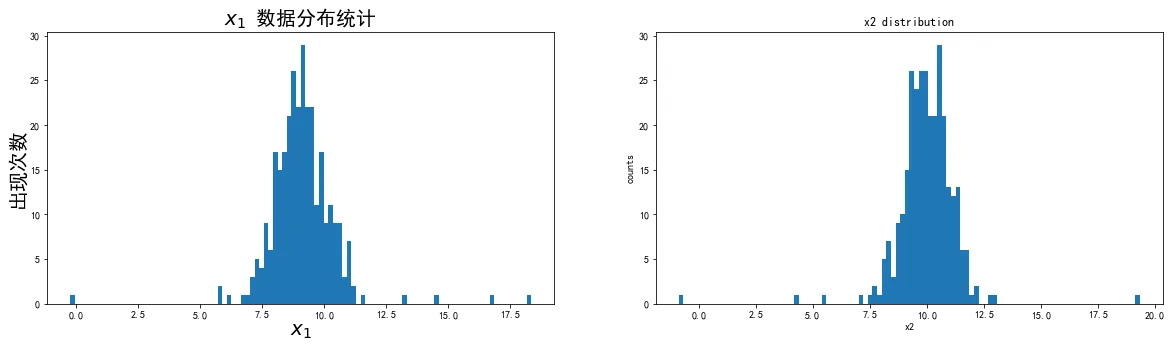
#define x1 and x2
x1 = data.loc[:,'x1']
x2 = data.loc[:,'x2']
# coding:utf-8
import matplotlib as mlp
font2 = {'family' : 'SimHei',
'weight' : 'normal',
'size' : 20,
}
mlp.rcParams['font.family'] = 'SimHei'
mlp.rcParams['axes.unicode_minus'] = False
fig2 = plt.figure(figsize=(20,5))
plt.subplot(121)
plt.hist(x1,bins=100)
plt.title('$x_1$ 数据分布统计',font2)
plt.xlabel('$x_1$',font2)
plt.ylabel('出现次数',font2)
plt.subplot(122)
plt.hist(x2,bins=100)
plt.title('x2 distribution')
plt.xlabel('x2')
plt.ylabel('counts')
plt.show()
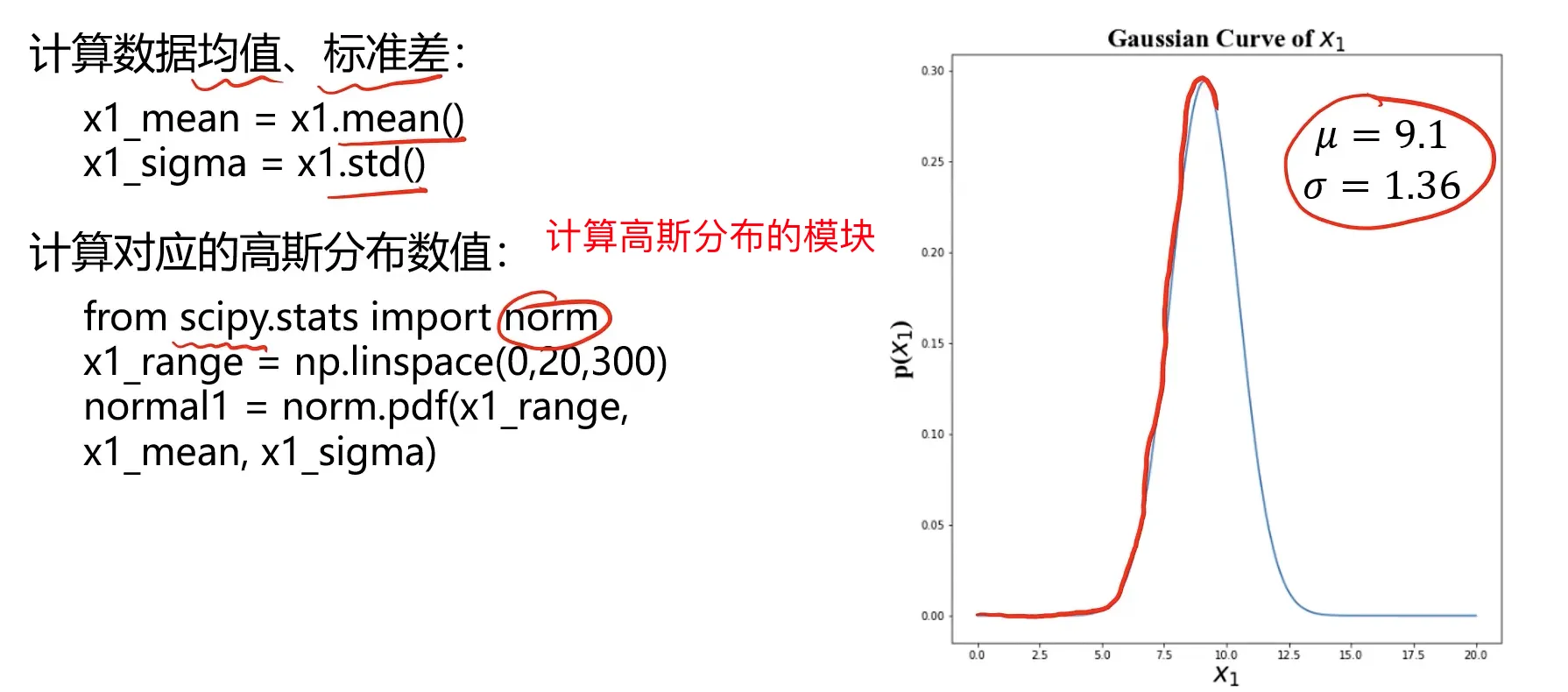
#calculate the mean and sigma of x1 and x2
x1_mean = x1.mean()
x1_sigma = x1.std()
x2_mean = x2.mean()
x2_sigma = x2.std()
print(x1_mean,x1_sigma,x2_mean,x2_sigma)
9.112225783931596 1.3559573758220915 9.997710507954398 1.30970711753864
#calculate the gaussian distribution p(x)
from scipy.stats import norm
x1_range = np.linspace(0,20,300)
x1_normal = norm.pdf(x1_range,x1_mean,x1_sigma)
x2_range = np.linspace(0,20,300)
x2_normal = norm.pdf(x2_range,x2_mean,x2_sigma)
#visualize the p(x)
fig2 = plt.figure(figsize=(20,5))
plt.subplot(121)
plt.plot(x1_range,x1_normal)
plt.title('normal p(x1)')
plt.subplot(122)
plt.plot(x2_range,x2_normal)
plt.title('normal p(x2)')
plt.show()

#establish the model and predict
from sklearn.covariance import EllipticEnvelope
ad_model = EllipticEnvelope(contamination=0.03)
ad_model.fit(data)
| EllipticEnvelope(assume_centered=False, contamination=0.03, random_state=None,store_precision=True, support_fraction=None) |
|---|
训练完毕后可以看出存在0.03的异常点
#make prediction
y_predict = ad_model.predict(data)
print(pd.value_counts(y_predict))
1 297
-1 10
dtype: int64
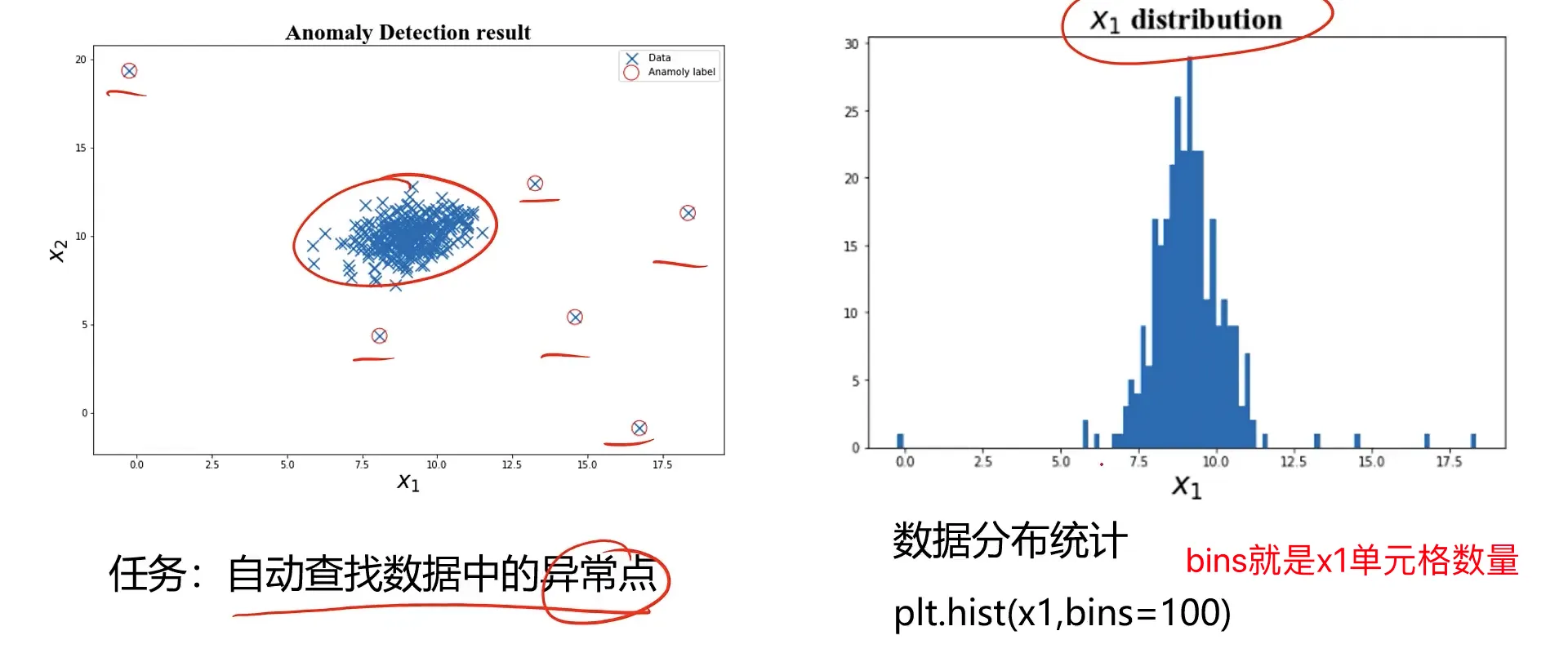
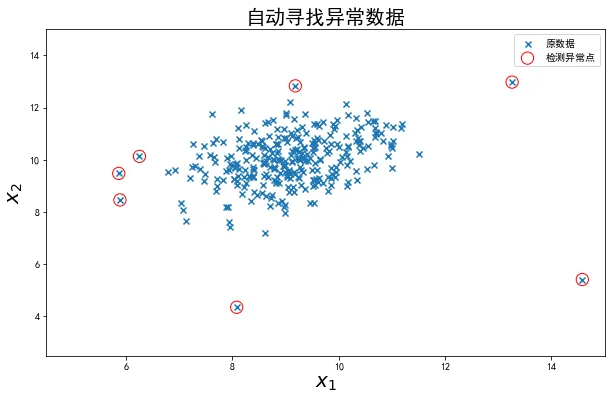
#visualize the result
fig4 = plt.figure(figsize=(10,6))
orginal_data=plt.scatter(data.loc[:,'x1'],data.loc[:,'x2'],marker='x')
anomaly_data=plt.scatter(data.loc[:,'x1'][y_predict==-1],data.loc[:,'x2'][y_predict==-1],marker='o',facecolor='none',edgecolor='red',s=150)
plt.title('自动寻找异常数据',font2)
plt.xlabel('$x_1$',font2)
plt.ylabel('$x_2$',font2)
plt.legend((orginal_data,anomaly_data),('原数据','检测异常点'))
plt.axis([4.5,15,2.5,15])
plt.show()
ad_model = EllipticEnvelope(contamination=0.02)
ad_model.fit(data)
y_predict = ad_model.predict(data)
#visualize the result
fig5 = plt.figure(figsize=(20,10))
orginal_data=plt.scatter(data.loc[:,'x1'],data.loc[:,'x2'],marker='x')
anomaly_data=plt.scatter(data.loc[:,'x1'][y_predict==-1],data.loc[:,'x2'][y_predict==-1],marker='o',facecolor='none',edgecolor='red',s=150)
plt.title('anomaly detection result')
plt.xlabel('x1')
plt.ylabel('x2')
plt.legend((orginal_data,anomaly_data),('original_data','anomaly_data'))
plt.show()
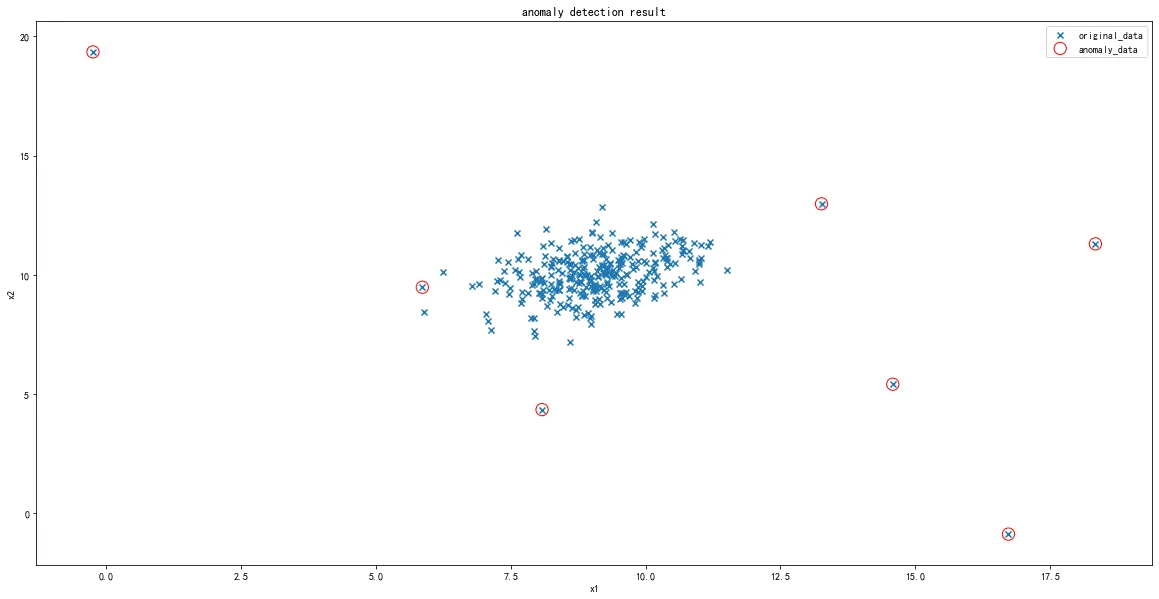
1、通过计算数据各维度对应的高斯分布概率密度函数,可用于寻找到数据中的异常点;
2、通过修改概率密度阈值contamination,可调整异常点检测的灵敏度;
上面的核心代码就是:
#x1:展示的数据 bins:横坐标划分了100个小格子
plt.hist(x1,bins = 100)
#计算数据均值,标准差
x1_mean = x1.mean()
x1_sigma = x1.std()
#计算对应的高斯分布数值
#linspacce(0,20,300):生成0~20的300个数据点
#pdf:概率密度函数点
from scipy.stats import norm
x1_range = np.linspacce(0,20,300)
normal1 = norm.pdf(x1_range,x1_mean,x1_sigma)
#可视化高斯分布曲线
plt.plot(x1_range,normal1)
#模型训练
from sklearn.covariance import EllipticEnvelope
clf = EllipticEnvelope()
clf.fit(data)
#可视化异常数据
anamoly_points = plt.scatter(data.loc[:,'x1'][y_predict == -1],data.loc[:,'x2'][y_predict == -1],marker = 'o',facecolor = 'none',edgecolor = 'red'.s = '250')
PCA实战
1、基于iris_data.csv数据,建立KNN模型实现数据分类(n_neighbors=3)
2、对数据进行标准化处理,选取一个维度可视化处理后的效果
3、进行与原数据等维度PCA,查看各主成分的方差比例
4、保留合适的主成分,可视化降维后的数据
5、基于降维后数据建立KNN模型,与原数据表现进行对比
先使用KNN进行分类:
#load data
import pandas as pd
import numpy as np
data = pd.read_csv('iris_data.csv')
data.head()
<table border="1" class="dataframe"> <thead> <tr style="text-align: right;"> <th></th> <th>sepal length</th> <th>sepal width</th> <th>petal length</th> <th>petal width</th> <th>target</th> <th>label</th> </tr> </thead> <tbody> <tr> <th>0</th> <td>5.1</td> <td>3.5</td> <td>1.4</td> <td>0.2</td> <td>Iris-setosa</td> <td>0</td> </tr> <tr> <th>1</th> <td>4.9</td> <td>3.0</td> <td>1.4</td> <td>0.2</td> <td>Iris-setosa</td> <td>0</td> </tr> <tr> <th>2</th> <td>4.7</td> <td>3.2</td> <td>1.3</td> <td>0.2</td> <td>Iris-setosa</td> <td>0</td> </tr> <tr> <th>3</th> <td>4.6</td> <td>3.1</td> <td>1.5</td> <td>0.2</td> <td>Iris-setosa</td> <td>0</td> </tr> <tr> <th>4</th> <td>5.0</td> <td>3.6</td> <td>1.4</td> <td>0.2</td> <td>Iris-setosa</td> <td>0</td> </tr> </tbody> </table>
#define X and y
X = data.drop(['target','label'],axis=1)
y = data.loc[:,'label']
y.head()
0 0
1 0
2 0
3 0
4 0
Name: label, dtype: int64
#establish knn model and calculate the accuracy
from sklearn.neighbors import KNeighborsClassifier
KNN = KNeighborsClassifier(n_neighbors=3)
KNN.fit(X,y)
y_predict = KNN.predict(X)
from sklearn.metrics import accuracy_score
accuracy = accuracy_score(y,y_predict)
print(accuracy)
0.96
使用KNN有0.96的准确率。
#数据标准化
from sklearn.preprocessing import StandardScaler
X_norm = StandardScaler().fit_transform(X)
print(X_norm)
[[-9.00681170e-01 1.03205722e+00 -1.34127240e+00 -1.31297673e+00]
[-1.14301691e+00 -1.24957601e-01 -1.34127240e+00 -1.31297673e+00]
[-1.38535265e+00 3.37848329e-01 -1.39813811e+00 -1.31297673e+00]
[-1.50652052e+00 1.06445364e-01 -1.28440670e+00 -1.31297673e+00]
[-1.02184904e+00 1.26346019e+00 -1.34127240e+00 -1.31297673e+00]
[-5.37177559e-01 1.95766909e+00 -1.17067529e+00 -1.05003079e+00]
[-1.50652052e+00 8.00654259e-01 -1.34127240e+00 -1.18150376e+00]
[-1.02184904e+00 8.00654259e-01 -1.28440670e+00 -1.31297673e+00]
[-1.74885626e+00 -3.56360566e-01 -1.34127240e+00 -1.31297673e+00]
[-1.14301691e+00 1.06445364e-01 -1.28440670e+00 -1.44444970e+00]
[-5.37177559e-01 1.49486315e+00 -1.28440670e+00 -1.31297673e+00]
[-1.26418478e+00 8.00654259e-01 -1.22754100e+00 -1.31297673e+00]
[-1.26418478e+00 -1.24957601e-01 -1.34127240e+00 -1.44444970e+00]
[-1.87002413e+00 -1.24957601e-01 -1.51186952e+00 -1.44444970e+00]
……
#calcualte the mean and sigma
x1_mean = X.loc[:,'sepal length'].mean()
x1_norm_mean = X_norm[:,0].mean()
x1_sigma = X.loc[:,'sepal length'].std()
x1_norm_sigma = X_norm[:,0].std()
print(x1_mean,x1_sigma,x1_norm_mean,x1_norm_sigma)
5.843333333333334 0.828066127977863 -4.736951571734001e-16 1.0
%matplotlib inline
from matplotlib import pyplot as plt
fig1 = plt.figure(figsize=(20,5))
plt.subplot(121)
plt.hist(X.loc[:,'sepal length'],bins=100)
plt.subplot(122)
plt.hist(X_norm[:,0],bins=100)
plt.show()
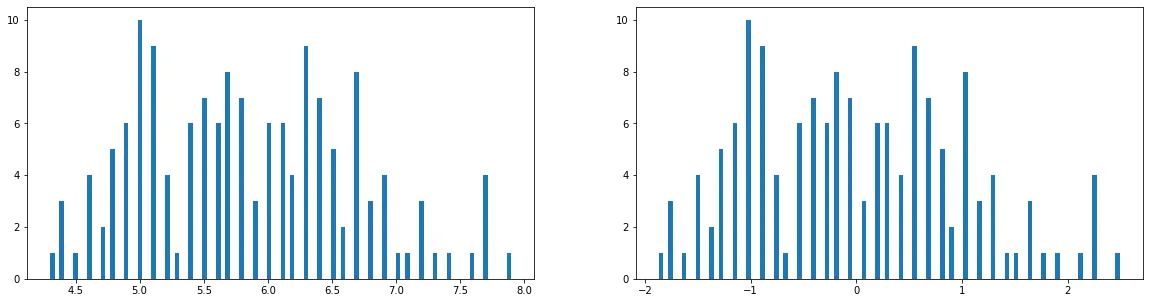
目前是150条数据,每条数据4个维度
print(X.shape)
(150, 4)
开始主成分分析(PCA)
#pca analysis
from sklearn.decomposition import PCA
pca = PCA(n_components=4)
X_pca = pca.fit_transform(X_norm)
#calculate the variance ratio of each principle components
var_ratio = pca.explained_variance_ratio_
print(var_ratio)
[0.72770452 0.23030523 0.03683832 0.00515193]
fig2 = plt.figure(figsize=(20,5))
plt.bar([1,2,3,4],var_ratio)
plt.xticks([1,2,3,4],['PC1','PC2','PC3','PC4'])
plt.ylabel('variance ratio of each PC')
plt.show()
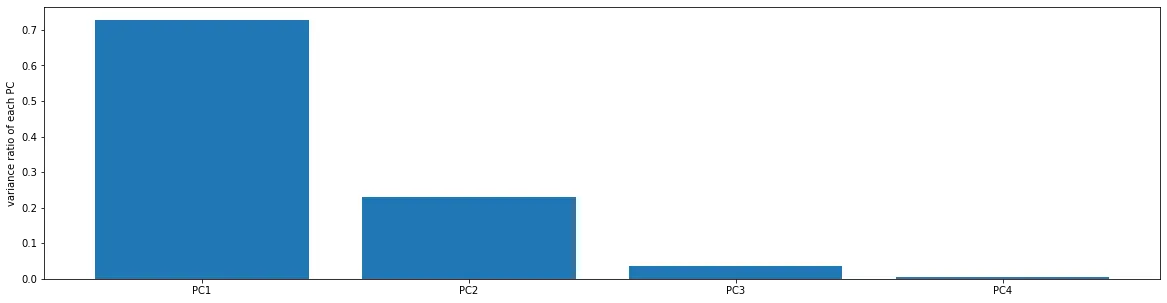
可以看到只有两个维度协方差比较大,所以数据维度可以降为2
pca = PCA(n_components=2)
X_pca = pca.fit_transform(X_norm)
X_pca.shape
(150, 2)
可以看到是150条数据,每条数据2个维度
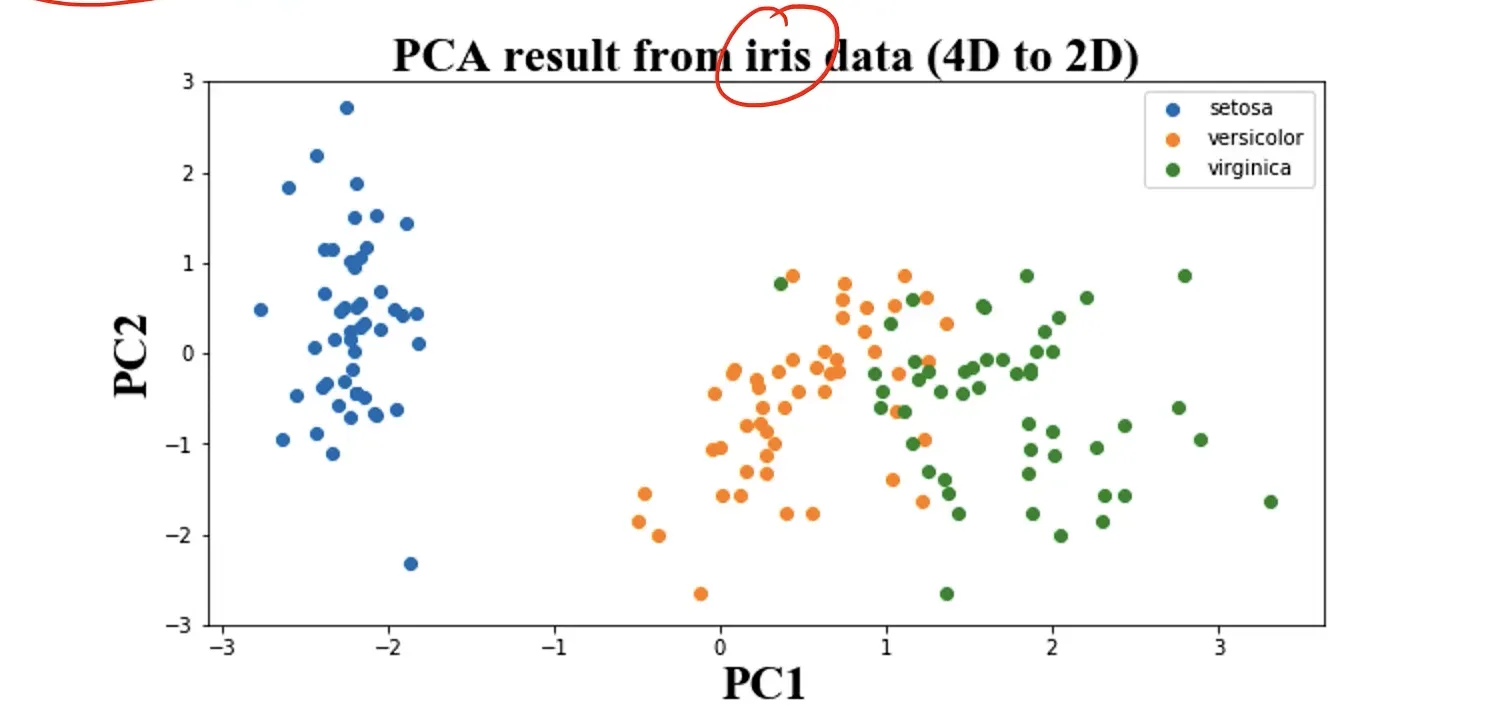
#visualize the PCA result
fig3 = plt.figure(figsize=(5,3))
setosa=plt.scatter(X_pca[:,0][y==0],X_pca[:,1][y==0])
versicolor=plt.scatter(X_pca[:,0][y==1],X_pca[:,1][y==1])
virginica=plt.scatter(X_pca[:,0][y==2],X_pca[:,1][y==2])
plt.legend((setosa,versicolor,virginica),('setosa','versicolor','virginica'))
plt.xlabel('PC1')
plt.ylabel('PC2')
plt.show()
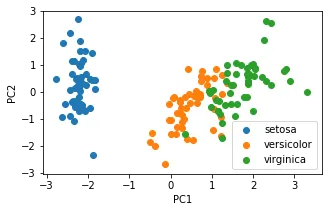
此时通过这个只有两个维度的数据建立KNN,与之前4个数据维度的KNN模型进行对比:
KNN = KNeighborsClassifier(n_neighbors=3)
KNN.fit(X_pca,y)
y_predict = KNN.predict(X_pca)
from sklearn.metrics import accuracy_score
accuracy = accuracy_score(y,y_predict)
print(accuracy)
0.9466666666666667
之前是0.96,现在是0.94,只有极少数数据没有得到正确分类,但是维度却降低了2个,这在处理大规模的数据的时候降维将是非常行之有效的手段。精简后的代码如下:
#数据标准化预处理
from sklearn.preprocessing import StandardScaler
X_norm = StandardScaler().fit_transfrom(X)
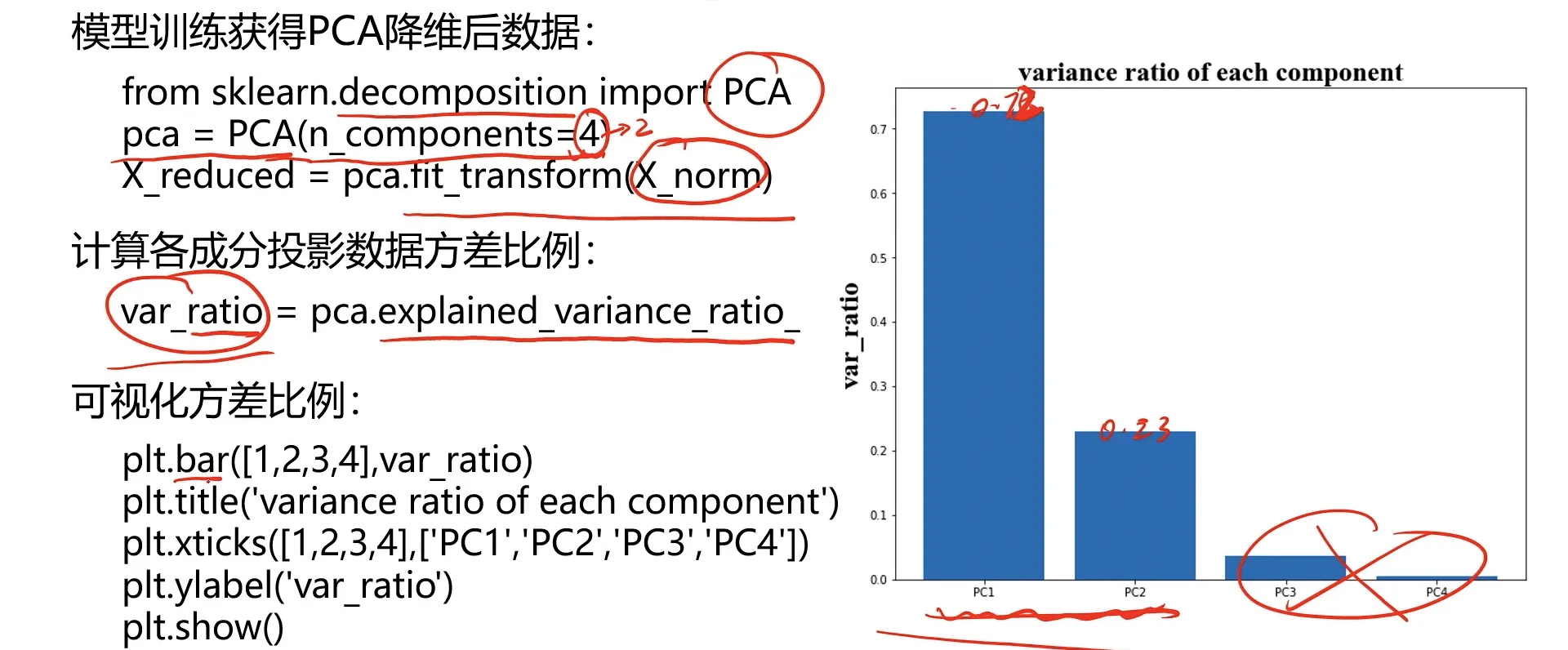
#模型训练获得PCA降维后数据
from sklearn.decomposition import PCA
pca = PCA(n_components = 4)
X_reduced = pca.fit_transform(X_norm)
#计算各成分投影数据方差比例
var_ratio = pca.explained_variance_ratio_
#可视化方差比例
#[1,2,3,4]:想画4个比例的
plt.bar([1,2,3,4],var_ratio)
plt.title('variance ratio of each component')
plt.xticks([1,2,3,4],['PC1','PC2','PC3','PC4'])
plt.ylabel('var_ratio')
plt.show()
#可视化PCA后数据
setosa = plt.scatter(X_reduced[:,0][y==0],X_reduced[:,1][y==0])
versicolor = plt.scatter(X_reduced[:,0][y==1],X_reduced[:,1][y==1])
virginica = plt.scatter(X_reduced[:,0][y==2],X_reduced[:,1][y==2])